Relación del CoV del SRAS con otros virus-ARN patógenos estudiada mediante patrones de tetranucleótidos
nov 22nd, 2003. En: SRAS. ![]()
En este momento el origen exacto de la causa del síndrome respiratorio agudo severo (SRAS) sigue siendo una incógnita. En este trabajo fue estudiada la relación de la secuencia genómica del SARS-CoV con 30 diferentes virus de ARN (ssRNA) monocatenario de varias familias, usando dos enfoques no estándares. Ambos enfoques se iniciaron con el perfilado vectorial del patrón V de uso del tetranucleótido para cada virus.
En el enfoque uno, se ideó una medida de distancia de un vector V, basado en el coeficiente de correlación, para construir el árbol filogenético por el algoritmo de unión de vecinos. En el enfoque de dos, se realizó un análisis multivariado de factores para derivar los patrones de uso del tetranucleótido incorporados. Estos patrones fueron utilizados posteriormente para clasificar los virus seleccionados.
Ambos enfoques rindieron resultados consistentes con la clasificación conocida de virus. También indicaron que el genoma de los virus ARN de la misma familia se ajustan a un patrón específico de uso. Basado en la correlación de los patrones de uso general de tetranucleótido, el Virus de la Gastroenteritis Transmisible (TGV) y el CoronaVirus felino (FCoV) son los más cercanos al SARS-CoV. Sorprendentemente, los virus ARN que no pasan por una etapa de ADN muestran una notable discriminación contra el CpG y UpA di-nucleótido (z = -77.31, -52.48 respectivamente) y selección para UpG y CpA (z = 65.79, 49.99 respectivamente). Se discuten posibles factores que influyen en estos sesgos.
El estudio del uso de la palabra genómica es un método eficaz para clasificar los virus ARN. La congruencia de los resultados de relación con la conocida clasificación, indica que existen señales filogenéticas en los patrones de uso de tetranucleótido, que es más prominente en el marco de lectura abierta de la replicasa.
Relationship of SARS-CoV to other pathogenic RNA viruses explored by tetranucleotide usage profiling. Yee Leng Yap, Xue Wu Zhang and Antoine Danchin. BMC Bioinformatics 2003, 4:43





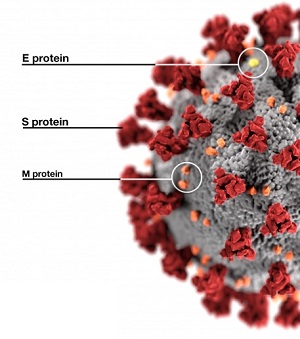
 Las infecciones provocadas por los coronavirus son comunes en todo el mundo y pueden afectar a personas y animales. En los humanos pueden causar el catarro común y la gastroenteritis en lactantes. Existen varios tipos de coronavirus y aunque por lo general ocasionan enfermedades leves o moderadas, también pueden estar implicados en procesos graves...
Las infecciones provocadas por los coronavirus son comunes en todo el mundo y pueden afectar a personas y animales. En los humanos pueden causar el catarro común y la gastroenteritis en lactantes. Existen varios tipos de coronavirus y aunque por lo general ocasionan enfermedades leves o moderadas, también pueden estar implicados en procesos graves... 



Comentar