Vigilancia genómica y secuenciación del SARS-CoV-2 en América del Sur
 Tras dos años de pandemia de COVID-19, los protocolos empleados para controlar la infección carecen de atención y análisis. En este artículo se presentan datos sobre depósitos de secuencias genómicas completas del SARS-CoV-2 en la base de datos de secuenciación GISAID.
Tras dos años de pandemia de COVID-19, los protocolos empleados para controlar la infección carecen de atención y análisis. En este artículo se presentan datos sobre depósitos de secuencias genómicas completas del SARS-CoV-2 en la base de datos de secuenciación GISAID.
GISAID es la Iniciativa mundial para intercambiar todos los datos sobre la gripe aviar, realizadas entre enero del 2021 y el 31 de mayo del 2022.
Se creó el perfil de distribución de las variantes del SARS-CoV-2 en América del Sur, en el que se destacaron la contribución y la influencia de cada variante a lo largo del tiempo.
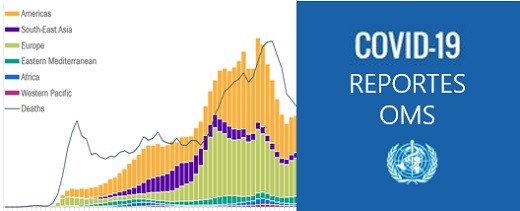
El monitoreo de las secuencias genómicas en GISAID ilustra la negligencia en el seguimiento de los pacientes infectados en América del Sur, así como las discrepancias entre el número de genomas completos depositados a lo largo de la pandemia por parte de los países desarrollados y los países en desarrollo. Mientras que Europa y América del Norte han depositado más de 9 millones de genomas en GISAID, África y América del Sur han aportado menos de 400 000 secuencias genómicas.
La vigilancia genómica es importante para detectar los primeros signos de alerta de virus nuevos en circulación, ayudar en el descubrimiento de nuevas variantes y controlar las pandemias.
Vea el artículo en:





 Las infecciones provocadas por los coronavirus son comunes en todo el mundo y pueden afectar a personas y animales. En los humanos pueden causar el catarro común y la gastroenteritis en lactantes. Existen varios tipos de coronavirus y aunque por lo general ocasionan enfermedades leves o moderadas, también pueden estar implicados en procesos graves...
Las infecciones provocadas por los coronavirus son comunes en todo el mundo y pueden afectar a personas y animales. En los humanos pueden causar el catarro común y la gastroenteritis en lactantes. Existen varios tipos de coronavirus y aunque por lo general ocasionan enfermedades leves o moderadas, también pueden estar implicados en procesos graves... 




Comentar