Coronavirus Genomics and Bioinformatics Analysis
ene 30th, 2017. En: NotiWeb. ![]()
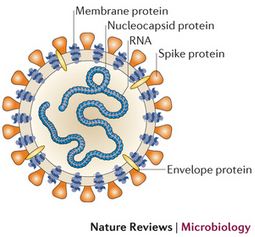
 El drástico aumento en el número de coronavirus descubiertos y de genomas de coronavirus que se han secuenciando, constituye una oportunidad sin precedentes para realizar análisis genómicos y bioinformáticos de esta familia de virus. Tradicionalmente, los virus se caracterizaron y clasificaron por cultivo, microscopía electrónica y estudios serológicos. Usando estos métodos fenotípicos, los coronavirus se definieron como virus envueltos de 120-160 nm de diámetro con una apariencia de corona.
El drástico aumento en el número de coronavirus descubiertos y de genomas de coronavirus que se han secuenciando, constituye una oportunidad sin precedentes para realizar análisis genómicos y bioinformáticos de esta familia de virus. Tradicionalmente, los virus se caracterizaron y clasificaron por cultivo, microscopía electrónica y estudios serológicos. Usando estos métodos fenotípicos, los coronavirus se definieron como virus envueltos de 120-160 nm de diámetro con una apariencia de corona.
La invención y los avances en tecnologías de amplificación de ácidos nucleicos, secuenciación automatizada de ADN y herramientas bioinformáticas en las últimas dos décadas, han revolucionado la caracterización y clasificación de todo tipo de agentes de enfermedades infecciosas. Utilizando métodos moleculares, los coronavirus se clasificaron como virus de ARN monocatenario de sentido positivo.Además, los resultados de la utilización de métodos filogenéticos para la clasificación también ayudó a definir los límites del grupo de la clasificación antigénica tradicional.
Los coronavirus poseen los genomas más grandes entre todos los virus de ARN conocidos (26,4 a 31,7 kb). Filogenéticamente, existen tres géneros: Alphacoronavirus, Betacoronavirus y Gammacoronavirus, más los subgrupos A, B, C y D del género Betacoronavirus. Recién se identificó un cuarto género emergente, Deltacoronavirus, que incluye el coronavirus bulbul HKU11, el coronavirus HKU12 y el coronavirus HKU13 de munia. El análisis molecular reveló que el momento del ancestro común más reciente del coronavirus relacionado con el SRAS humano/civeta fue 1999-2002.
Según este estudio, la recombinación en los coronavirus fue más notable entre las diferentes cepas del virus de la hepatitis murina (MHV), entre diferentes cepas del virus de la bronquitis infecciosa, entre MHV y el coronavirus bovino, entre el coronavirus felino (FCoV) tipo I y el coronavirus canino generando FCoV tipo II, y entre tres genotipos de coronavirus humano HKU1 (HCoV-HKU1).
Bajo este nuevo sistema de clasificación, los coronavirus de los murciélagos dominan los géneros Alphacoronavirus y Betacoronavirus y los coronavirus de los pájaros dominan los géneros Gammacoronavirus y Deltacoronavirus. Esta enorme diversidad de coronavirus en murciélagos y aves los ha convertido en excelentes reservas genéticas para coronavirus en estos cuatro géneros.
Si bien este estudio se desarrolló y publicó antes de la aparición del coronavirus del MERS, proponemos su lectura por los interesantes hallazgos en materia de filogenética que aporta al conocimiento de este género.
Woo PCY, Huang Y, Lau SKP, Yuen K-Y. Coronavirus Genomics and Bioinformatics Analysis. Viruses. 2010;2(8):1804-1820. doi:10.3390/v2081803.





 Las infecciones provocadas por los coronavirus son comunes en todo el mundo y pueden afectar a personas y animales. En los humanos pueden causar el catarro común y la gastroenteritis en lactantes. Existen varios tipos de coronavirus y aunque por lo general ocasionan enfermedades leves o moderadas, también pueden estar implicados en procesos graves...
Las infecciones provocadas por los coronavirus son comunes en todo el mundo y pueden afectar a personas y animales. En los humanos pueden causar el catarro común y la gastroenteritis en lactantes. Existen varios tipos de coronavirus y aunque por lo general ocasionan enfermedades leves o moderadas, también pueden estar implicados en procesos graves... 


Comentar